糖蛋白的糖基化位点和寡糖的异质性的快速测定
当前位置:首页 >> 仪器仪表技术文章
糖蛋白的糖基化位点和寡糖的异质性的快速测定
摘要: 采用链霉蛋白酶进行酶解可以避免传统胰蛋白酶酶解法的一些不足。此外,与GlycoX程序结合后,该方法具有适用于高通量大规模的糖蛋白质组学研究的潜力。
糖基化是原核蛋白中最常见的翻译后修饰形式之一[1]。糖基化位点和寡糖异质性的测定对于理解糖蛋白特殊的生物学作用是至关重要的。据估计至少有50%的人源蛋白经过糖基化修饰[2]。最主要的两类糖基化修饰方式是N端糖基化和O端糖基化。N端多糖经氨基连接在天冬酰胺残基上,具有特定序列N-X-(S或者T)。这里X可以是除脯氨酸外的任意一种氨基酸。N端连接的聚糖具有由两个N-乙酰氨基葡萄糖(GlcNAc)和三个甘露糖(Man)残基形成的单核。O端的聚糖连接到丝氨酸或苏氨酸上,但没有单一共同的单核或特定的蛋白序列。
由于糖蛋白的高度复杂性,糖基化位点分析具有极大的挑战性。最近有一些关于糖基化位点分析的报道[3-11]。常用方法包括以下步骤(见图1a):专一蛋白酶的酶解(通常是胰蛋白酶)、糖肽的富集(通常采用液相色谱或亲和色谱)和糖肽的质谱分析[7-11]。有时也可通过糖肽的去糖基化获得丢失的聚糖信息[7,11]。然而许多糖蛋白不易被胰蛋白酶酶解[12,13]。此外,由于糖基化会造成胰蛋白酶酶切位点的丢失,因此酶解产生的糖肽太大而不适于质谱分析[9]。基于上述原因,目前获得的糖基化信息通常是不完全的。
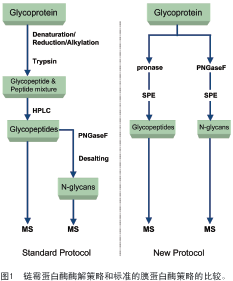
作者所在的实验室中发展了一种独特的能够同时测定糖蛋白中N端糖基化位点和寡糖异质性的方法(见图 1b)[4]。利用该方法,糖蛋白被非专一性的蛋白酶酶解。通过采用高活性的蛋白酶混合物,即链霉蛋白酶,形成大量较短的糖肽(含2~8个残基)。糖蛋白的非糖基化部分被酶解成氨基酸和二肽,而糖基化部分被寡糖保护从而抑制了蛋白酶的活性。采用装有多孔石墨化碳(PGC)的固相萃取(SPE)柱可以很容易地将产生的糖肽与氨基酸和盐分离。同时,糖蛋白用 N-糖酰胺酶F(PNGase F)处理。PNGase F能够从糖蛋白中专一地切割N端糖基。然而该操作仅对于具有几个糖基化位点的较大糖蛋白是必要的,较小的糖蛋白不需要单独地测定寡糖的组成。在任何情况下释放的聚糖可采用装有PGC的SPE柱纯化。纯化的糖肽和聚糖可采用基质辅助激光解吸离子化质谱(MALDI-MS)和高精度的傅里叶变换离子回旋共振质谱(FT-ICR-MS)进行分析。采用后者可以完成对任一糖基化位点的确认和聚糖异质性分析。
1 链霉蛋白酶酶解
先将约10 nmol的糖蛋白溶解在0.1 mol/L Tris 缓冲液(pH 7.5)中,再用约10个单位的链霉蛋白酶处理。上述溶液在37 ℃放置36~48 h。同时,采用PNGase F在37 ℃ 条件下酶解糖蛋白12 h,释放 N端连接的聚糖。采用装有PGC的SPE柱纯化由非专一蛋白质水解产生的糖肽和由PNGase F释放的寡糖。将装有PGC的SPE柱依次用水和含有0.05%(v/v)三氟乙酸(TFA)的80%乙腈(ACN)/水(v/v)冲洗。再将酶解的糖蛋白或寡糖溶液上样到PGC柱上。随后用去离子水以1 mL/min的流速冲洗,去除盐和缓冲液。最后分别用含有0.05%TFA的10%ACN、20%ACN和40%ACN洗脱糖肽和多糖。在进行MALDI分析前,将每一次洗脱液分别收集并真空浓缩。
2 质谱
质谱采用配备7.0 特斯拉磁场和用于离子化的脉冲掺钕钇铝石榴石(Nd:YAG)激光(355 nm)的外源HiResMALDI(IonSpec Corp., Irvine, CA)。采用溶于5 mg/100 μL乙醇的2,5-二羟基安息香酸(DHB)作基体;采用饱和的氯化钠(或者氯化钾)-甲醇溶液作掺杂剂,以产生糖肽和寡糖的碱金属相关的准分子离子。将1 μL糖肽/寡糖溶液和1 μL的基体溶液依次滴加到MALDI靶体。样品在空气流中干燥后进行质谱分析。示波器 | 电阻计 | 可燃气体检测仪 | 粒子计数器 | 传送器 | 钩表 | PH计 | 万能钳 | 温湿度仪 | 溶氧计 | 试验机 | 温度记录仪 | 盐度计
3 糖基化位点的表征
为了验证该实验策略,对一个具有单一糖基化位点(60N)和已知寡糖结构的模式糖蛋白核糖核酸酶B进行了表征[14]。由链霉蛋白酶酶解后分离出的糖肽的正离子模式质谱图如图 2a所示。该谱图是在没有经过高效液相色谱(HPLC)分离而只采用最简单的样品预处理后获得的。由于被酶解为氨基酸,大的非糖肽碎片并没有出现。观察到的大于m/z 1 000的谱峰对应的是糖肽,这是因为寡糖配基的质量单位至少为800(GlcNAc2Man3)。在谱图中出现了两个糖肽系列(空心和实心的方块分别代表系列1和系列2),分子质量相差162 Da(1个甘露糖残基)。样品中添加NaCl可获得更多的信息。掺杂剂能增强系列1中5个峰的相对强度,这与钠离子的加成物相关(图 2a)。另一系列是质子化样品,其强度随着NaCl的加入而降低。
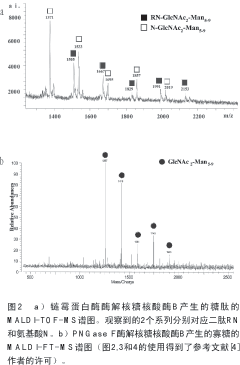
为了鉴定图 2a中的多肽碎片,采用PNGase F将多糖从糖蛋白中释放出来,并用MALDL-MS进行分析(图 2b)。图 2a中观察到的5个多糖峰代表5个糖肽,其信号分别相应于组成从GlcNAc- 2Man5到 GlcNAc2Man9的高甘露糖寡糖与钠离子的加成物。相邻谱峰的质量差是162 Da,相当于一个甘露糖残基。图 2b中的5个多糖的信号相对强度与图 2a中5个糖肽的一致。
通过从糖肽质量中减去观察到的多糖质量可以对多肽碎片进行鉴定。例如,图 2a系列2中的第一个峰(m/z 1 505.456)对应的是带有多糖GlcNAc-2Man5(m/z 1 257.420)的糖肽。肽段的质量可以由观察到的糖肽质量1 504.45 Da([M+H]+减去H+)减去观察到的多糖质量1 216.42([M+Na]+ 减去 Na+和H2O)获得。多肽的质量288.12 Da相当于糖基化位点在60N的二肽精氨酸-天冬氨酸(RN)(理论质量288.15 Da)。理论质量与实验质量的差异通常小于20 ppm,因此可以断定系列2中的糖肽包含带有从GlcNAc2Man5 到 GlcNAc2Man9的高甘露糖寡糖的二肽RN。系列1中质量最小的峰(m/ z 1 371.39)对应着带有寡糖GlcNAc2-Man5的糖肽(m/z 1 257.42)。132.07 Da的肽段质量对应的是天冬氨酸(理论质量132.05 Da),因此系列1中的糖肽包含1个单个N端残基。为了证明可根据离子的相对丰度进行定量分析,图 3给出了每个多糖的相对丰度测定结果。因为这些多糖的结构、大小和离子化效率相似,因此可以计算出糖蛋白中寡糖的相对丰度[15]。
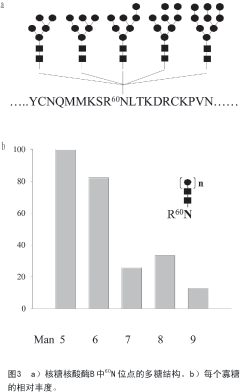
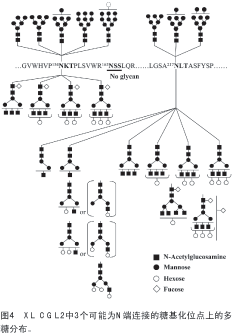
根据上述策略,可以确定从简单到复杂的糖蛋白中的专一糖基化位点,并获得内在的位点异质性信息。通过对模式蛋白如核糖核酸酶B和鸡卵清蛋白(含有两个潜在的位点,其中一个由计算得出)的分析,验证了结果的准确性。对于更复杂的含有未知糖基化位点的糖蛋白如来源于有爪蟾蜍(XL)和非洲蟾蜍(X)卵子的脑皮层颗粒凝集素(CGL1和CGL2)也进行了研究。分布在XL CGL2中N端糖基化位点上的多糖如图 4所示。采用该策略,也可对更复杂的糖蛋白如包括了7个潜在的未知糖基化位点的葡萄糖氧化酶进行分析。
4 GlycoX程序
作者的实验室还开发了一个新的用来帮助分析实验数据的电脑程序??GlycoX[16]。这个软件是在MATLAB基础上发展的,需要把质谱图以ASCII文档形式输入。它是利用已知蛋白序列的精确的糖肽和多糖质量来判断N端和O端连接的糖基化位点和微小的异质性。该程序能自动分析由非专一性或者低专一性蛋白酶水解(例如:链霉蛋白酶E或蛋白酶K)得到的糖肽质谱图。它具有三个主要功能:糖基化位点的搜索功能,用来判断糖蛋白的糖基化位点以及寡糖的异质性;寡糖计算功能,用来判断寡糖的组成是N端连接、O端连接还是经化学修饰的寡糖;自动同位素过滤功能,用来从糖肽、肽和多糖质谱图中选择单一的同位素峰。GlycoX流程图如图 5所示。
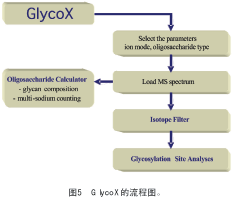
将质量强度(M/I)表(以ASCII格式保存)和从SWISS-PROT/TrEMBL数据库(FASTA格式,以文本文件保存)中获得的对应的糖蛋白的序列输入到GlycoX程序中。为了确定糖基化位点和对应的多糖,对经同位素过滤的质量进行分析,从而确定它们和哪种多糖和肽段质量对应。通过比较从单肽到用户定义的最大长度的肽段序列可以判断肽段的组成。利用该程序,也可以搜索匹配的分析量来判断多糖的质量。此外,也可以利用光谱来确认多糖的质量。对复杂的糖基化位点的分析,推荐采用多糖谱图对比的方法。
采用链霉蛋白酶酶解可以避免常用的胰蛋白酶酶解的一些缺点。此外,GlycoX程序具有适用于高通量大规模的糖蛋白质组学研究的潜力。
更多相关信息
分类点击排行
- 1500米防水温度记录仪MX2204 HOBO TidbiT MX Tempe 5000
- HOBO无线蓝牙水下温度记录仪MX2203 TidbiT MX
- MX2201新款带蓝牙水下温度记录仪Pendant MX Temp
- 防火报警智能热像Fotric816/Fotric826
- 工程师通用热像仪Fotric 222s/Fotric 222s-1/Fotric 222s-2
- HOBO MX100蓝牙温度记录仪防水等级IP67
- 可程式噪音计TES-1352S带记录可插SD卡
- 数据记录型照度光量子计TES-1339P
- 便携一体式温湿度大气压力计TES-1160/TES-1161
- 高精度水位计MX2001-04-s带无线数据传输


