以每次一个分子的方式研究整个基因组
当前位置:首页 >> 仪器仪表技术文章
以每次一个分子的方式研究整个基因组
摘要:基因组学新近的进展为生物学的发现创造了巨大的机遇,抓住这个机遇需要新颖的试验设计以及高通量的数据收集和分析系统。随着基因分析系统的灵活性和综合能力的增强,能够进行单细胞和单分子检测、适用于整个基因组、并且能够高通量的产生和处理巨大的数据平台的构建成为一种重要的趋势。由Wisconsin-Madison, WI大学构建Optical Mapping(可见图谱)系统便是这些新颖的平台之一,它能够以每次一个分子的方式研究整个基因组。
基因组学新近的进展为生物学的发现创造了一个巨大的充满机遇的舞台。抓住这个机遇需要新颖的试验设计、高通量的数据收集和分析系统以及富有创造性的方法去解决以前难以理解的问题。随着基因分析系统的灵活性及综合能力的增强,能够进行单细胞和单分子检测、适用于整个基因组、并且能够高通量的产生巨大的数据平台的构建正在成为一种重要的趋势。Optical Mapping(可见图谱)系统(University of Wisconsin-Madison, WI)作为这类新颖平台的一例,能够以每次一个分子的方式研究整个基因组。 电阻计 | 红外线测温仪 | 红外线温度计 | 频谱分析仪 | 钩表 | 多功能测试仪 | 电容表 | 记录仪 | 色度计 | 剥线钳 | 电压计 | 浊度计 | 温湿度记录仪
Optical Mapping 系统通过收集单个分子的图谱形成整个基因组的物理图谱。而这些物理图谱在基因组中能够反映特殊DNA 序列的位置以及它们之间的物理距离。在插入、删除、倒置、复制、颠换和重排方面,它们所进行的表征和比较对基因组非常有用[1]。通过创造性地结合DNA 分子的预处理和操作、表面化学、高速数据收集成像以及一系列的选择性处理过程,OpticalMapping 便产生出物理图谱,这些物理图谱的特征便是能为基因组学长期存在的问题提供新的解决办法。例如,Optical Mapping 的单分子检测功能使得它可以对不同种类的群体进行基因水平上的分析,比如实性瘤(solid tumor)内或者在细菌菌落里的分子的检测。从细胞中提取出的DNA 分子可直接成像,而不需要扩增和亚克隆步骤。因此基因的每一部位都可经Optical Mapping直接观测,使得这一系统成为一个真正的全基因方法。就此而言,Optical Mapping 系统与那些建立在整个染色体上进行基因比较的低分辨率的细胞遗传方法具有相似之处。但是,OpticalMapping 的分辨率可以达到几万个碱基,这使得该方法适合于低分辨方法辨基因型方法未曾被探索过的领域的研究。分辨率在几千个碱基的全基因分析方法开启了以前难以解决的基因变异问题之门,例如:对中等到大的片段插入和缺失的检测和翻译。更重要的是,通过Optical Mapping 检测基因组多态性不需要预先知道诸如基因的类型、位置等信息,使得这一方法成为名副其实的“发现型方法”。最后,Optical Mapping系统的通量相当于在90min 内收集代表一个人基因组的数据,如此大的通量也使得对于大范围群体的研究成为可能。在那种条件下,通过疾病敏感性和药物响应性基因座位鉴定的相关研究,整个人类基因组的全部变异都能被直接区分。下文中,作者会通过详细地描述Optical Mapping 系统,列举出该技术应用的典型实例,并把该技术置于现有基因分析平台之中来详细阐述Optical Mapping 的特点。
Optical Mapping 系统通过收集单个分子的图谱形成整个基因组的物理图谱。而这些物理图谱在基因组中能够反映特殊DNA 序列的位置以及它们之间的物理距离。在插入、删除、倒置、复制、颠换和重排方面,它们所进行的表征和比较对基因组非常有用[1]。通过创造性地结合DNA 分子的预处理和操作、表面化学、高速数据收集成像以及一系列的选择性处理过程,OpticalMapping 便产生出物理图谱,这些物理图谱的特征便是能为基因组学长期存在的问题提供新的解决办法。例如,Optical Mapping 的单分子检测功能使得它可以对不同种类的群体进行基因水平上的分析,比如实性瘤(solid tumor)内或者在细菌菌落里的分子的检测。从细胞中提取出的DNA 分子可直接成像,而不需要扩增和亚克隆步骤。因此基因的每一部位都可经Optical Mapping直接观测,使得这一系统成为一个真正的全基因方法。就此而言,Optical Mapping 系统与那些建立在整个染色体上进行基因比较的低分辨率的细胞遗传方法具有相似之处。但是,OpticalMapping 的分辨率可以达到几万个碱基,这使得该方法适合于低分辨方法辨基因型方法未曾被探索过的领域的研究。分辨率在几千个碱基的全基因分析方法开启了以前难以解决的基因变异问题之门,例如:对中等到大的片段插入和缺失的检测和翻译。更重要的是,通过Optical Mapping 检测基因组多态性不需要预先知道诸如基因的类型、位置等信息,使得这一方法成为名副其实的“发现型方法”。最后,Optical Mapping系统的通量相当于在90min 内收集代表一个人基因组的数据,如此大的通量也使得对于大范围群体的研究成为可能。在那种条件下,通过疾病敏感性和药物响应性基因座位鉴定的相关研究,整个人类基因组的全部变异都能被直接区分。下文中,作者会通过详细地描述Optical Mapping 系统,列举出该技术应用的典型实例,并把该技术置于现有基因分析平台之中来详细阐述Optical Mapping 的特点。
1 实验
在Optical Mapping 中,大的基因组DNA分子(300-3000kb 碱基)直接从细胞中提取、延伸,并通过结合微流控芯片技术[2]和带正电的玻璃表面与带负电的DNA 骨架之间的静电相互作用使其固定在被特殊处理的玻璃表面上(图1)。这种固定化技术相当精妙,以至限制性内切酶仍很容易接近DNA分子,而且产生的限制型片段也能得到很强的保留。可切割6000~45000 个碱基对的限制性内切酶能够用于制作高分辨率的物理图谱。荧光染料YOYO-1能够标记表面上的DNA骨架,其形成的单分子图谱的纳米阵列可通过荧光显微镜进行观测。

不管亚微观的DNA 分子的实际宽度是多少,荧光显微镜的数值放大功能使得DNA 分子的宽度有一个明显的增加,这样,被拉长的单链DNA 分子可很容易地通过标准的63×物镜观测到。大的、被拉长的DNA 分子的性质与拉长的弹簧相似,当它们紧紧黏附在限制性内切酶上,只有分子的末端保持松弛状态,各个分子之间便形成可以识别的1~2μm 的间隙。与大多数的荧光系统通过添加探针分子进行标记不同,Optical Mapping 以分子间荧光的消失作为标记。添加的探针分子通常会在标签周围产生模糊图案(由于传统的探针非特异杂交),产生相应的噪音。而这种方法不仅可以避免噪声的产生,也省去了后续的探针去除步骤。由于限制性内切酶具有特异性好和可预测的特点,Optical Mapping 可以保证物理图谱形成过程简单、准确。
快速数据收集通过一个可测定纳米阵列OpticalMapping 的面积的成像系统实现。与其他物理图谱方法不同,Optical Mapping 不需要知道碎片大小和序列信息。通过测量已知大小的DNA分子总的荧光强度,基于荧光强度随碱基对的变化即可以得到Optical Mapping 的碎片大小。尽管DNA 分子轮廓长度发生了变化,荧光强度仍保持一致,使得碎片大小测量仍很准确。由于D N A 碎片通过静电作用被固定在玻璃表面上,因此可实现单个分子限制性图谱的绘制。
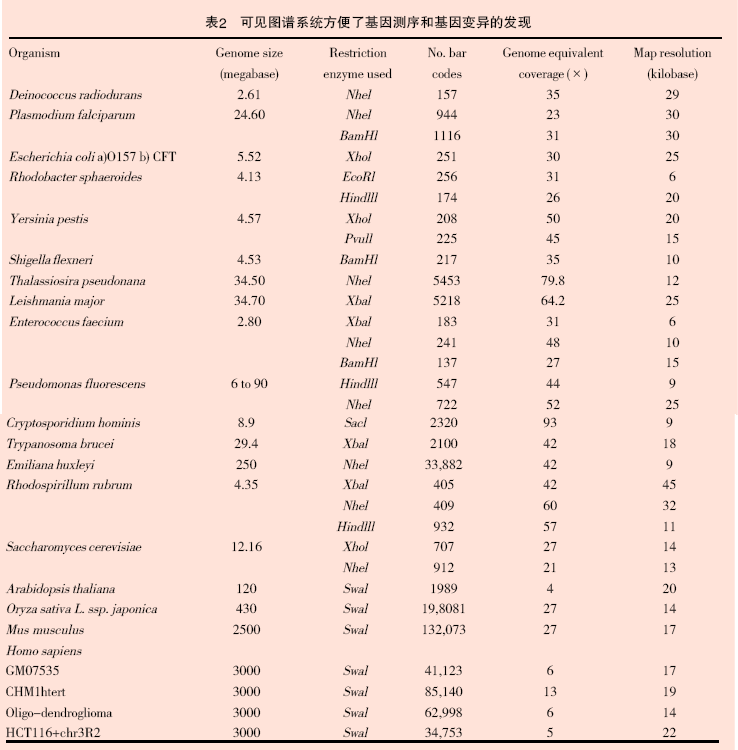
所有的单分子图谱组合在一起形成一个包含整个染色体的图谱,这一过程类似于序列的组装。与其他物理图谱不同,全基因Optical Mapping,又称“一致”图谱,综合了成千上万的单分子图谱。“一致”图谱上一个典型的基因座位覆盖了10~70 个单分子图谱。这么大的覆盖率使得Optical Mapping 比起其他的物理图谱技术有更高的准确性[3-9]。由于每个DNA 分子都能产生一系列离散的数据(限制性酶切碎片图谱),因而任何一个(或者每一个)DNA 分子产生的数据相当于琼脂糖凝胶上的一条完整的带或者微阵列中的一个点(见表1 )。这样,Optical Mapping 系统在一块芯片上产生一系列大量的类似数据,该方法的准确性和精确性已被上千次的实验所证实。
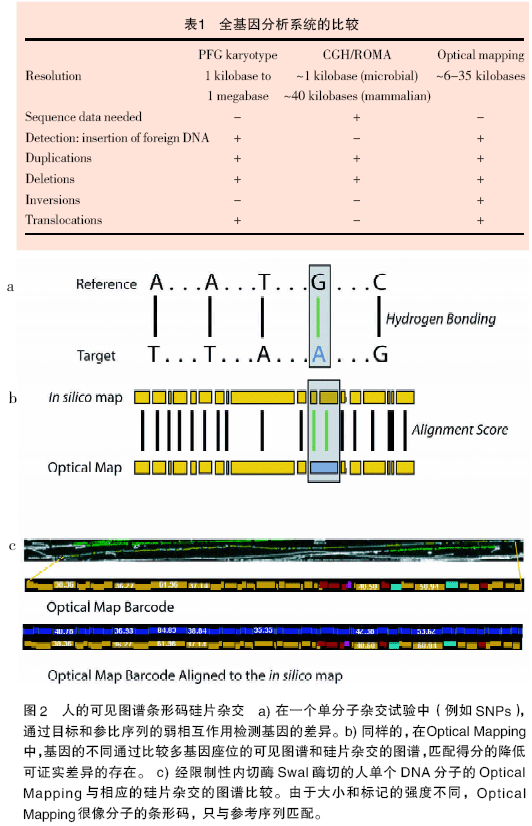
根据分析的目的,“一致”图谱和单分子Optical Mapping 都可以通过硅片杂交定位到相应的序列。就像扫描条形码鉴定物质一样(图2),通过扫描限制性碎片形成的Optical Mapping 图案(而不是在硅片上的图谱的图案)鉴定图谱相应的序列,进而可指认其在基因上的位置。在微阵列杂交试验中,碱基之间的氢键反映相应的目标和参考序列的匹配性,荧光强度的偏差表征多态性区域。同样,在硅片杂交中限制性碎片之间的得分可以反映Optical Mapping 和硅片图谱匹配的程度,而得分减少可表征多态性区域。硅片杂交的参数能被调整,从而使Optical Mapping 的条形码与序列准确地对应起来,但是也能很自由地检测染色体的区别。
快速数据收集通过一个可测定纳米阵列OpticalMapping 的面积的成像系统实现。与其他物理图谱方法不同,Optical Mapping 不需要知道碎片大小和序列信息。通过测量已知大小的DNA分子总的荧光强度,基于荧光强度随碱基对的变化即可以得到Optical Mapping 的碎片大小。尽管DNA 分子轮廓长度发生了变化,荧光强度仍保持一致,使得碎片大小测量仍很准确。由于D N A 碎片通过静电作用被固定在玻璃表面上,因此可实现单个分子限制性图谱的绘制。
所有的单分子图谱组合在一起形成一个包含整个染色体的图谱,这一过程类似于序列的组装。与其他物理图谱不同,全基因Optical Mapping,又称“一致”图谱,综合了成千上万的单分子图谱。“一致”图谱上一个典型的基因座位覆盖了10~70 个单分子图谱。这么大的覆盖率使得Optical Mapping 比起其他的物理图谱技术有更高的准确性[3-9]。由于每个DNA 分子都能产生一系列离散的数据(限制性酶切碎片图谱),因而任何一个(或者每一个)DNA 分子产生的数据相当于琼脂糖凝胶上的一条完整的带或者微阵列中的一个点(见表1 )。这样,Optical Mapping 系统在一块芯片上产生一系列大量的类似数据,该方法的准确性和精确性已被上千次的实验所证实。
根据分析的目的,“一致”图谱和单分子Optical Mapping 都可以通过硅片杂交定位到相应的序列。就像扫描条形码鉴定物质一样(图2),通过扫描限制性碎片形成的Optical Mapping 图案(而不是在硅片上的图谱的图案)鉴定图谱相应的序列,进而可指认其在基因上的位置。在微阵列杂交试验中,碱基之间的氢键反映相应的目标和参考序列的匹配性,荧光强度的偏差表征多态性区域。同样,在硅片杂交中限制性碎片之间的得分可以反映Optical Mapping 和硅片图谱匹配的程度,而得分减少可表征多态性区域。硅片杂交的参数能被调整,从而使Optical Mapping 的条形码与序列准确地对应起来,但是也能很自由地检测染色体的区别。
2 应用
2.1 从源头进行基因组的构建
2.1 从源头进行基因组的构建
基因组测序依赖于精确的物理图谱来形成和验证序列数据[1]。传统的全基因物理图谱通过酶切代表性的人工细菌染色体文库(BAC),以及综合产生的限制性片段组合成对应的基因组序列。现有技术如“高信息量指纹图谱“(HICF)[10]极大地改进了以BAC 为基础的物理图谱的分辨率和准确度。可是以BAC 为基础的技术由于对桥连大的局部重复的DNA 分子有困难,需要深入的染色体文库构建,并且单个样品需要很大的劳动强度,因而使得这一技术的优势被部分掩盖。
Optical Mapping 不仅不存在上述问题,而且极大地简化了物理图谱的形成过程。HICF 形成的指纹图谱没有碎片的序列信息,只是在一个克隆过程中形成的大量的无序碎片。大的DNA 分子被酶切成规则的并具有丰富信息的限制性碎片,而并非简单的指纹图谱。
Optical Mapping 除了用于表2 给出的基因组研究外,也被用于辅助其他测序过程,及对一些生物的基因水平研究(耐辐射球菌、恶性疟原虫、大肠杆菌、白色念珠菌、红螺菌、鼠疫杆菌、弗氏志贺氏菌、海链藻、利什曼原虫等)[3,6,7,11-15]。这些研究证实了通过Optical Mapping 系统构建物理图谱的精确性和稳定性。
Optical Mapping 不仅不存在上述问题,而且极大地简化了物理图谱的形成过程。HICF 形成的指纹图谱没有碎片的序列信息,只是在一个克隆过程中形成的大量的无序碎片。大的DNA 分子被酶切成规则的并具有丰富信息的限制性碎片,而并非简单的指纹图谱。
Optical Mapping 除了用于表2 给出的基因组研究外,也被用于辅助其他测序过程,及对一些生物的基因水平研究(耐辐射球菌、恶性疟原虫、大肠杆菌、白色念珠菌、红螺菌、鼠疫杆菌、弗氏志贺氏菌、海链藻、利什曼原虫等)[3,6,7,11-15]。这些研究证实了通过Optical Mapping 系统构建物理图谱的精确性和稳定性。
2.2 发现插入缺失的比较基因组学研究
Optical Mapping 系统不需要预先知道基因组的序列或结构,因此能很方便地比较相关的种类和分析来自同一物种中不同成员的多形态基因组。最近, 未经测序的Shigella flexneri Y血清型菌株的Optical Mapping 被构建出来,并与已测序的Shigella菌株的硅片图谱进行比较。在未测序的Y 血清型菌株中发现了明显的删除现象,包括葡糖转移酶gtrII、gtrAI 和gtrBI,这些转移酶可能包含在血型的变化过程中[13]。近来的研究强调基因改变过程中的插入和删除作用( i n d e l s )[16] 。用于检测indels 的完善系统还处于研究的初期,而推测indels 在人类基因组学中的作用和影响基于高分辨率或者低分辨率的基因组学方法。Optical Mapping 对人类基因组学的重要贡献便是发现了indels以及其他相似的现象如倒置、置换、复制和重排等,而其他高通量的基因组学平台很难达到如此高的分辨率。
2.3 基因组异常的完善检测方法
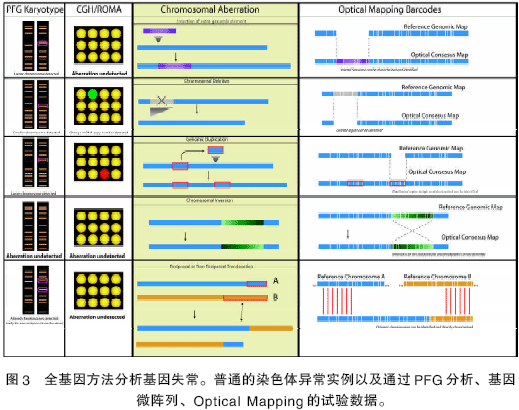
比较基因组杂交技术(CGH)和代表性的低聚核苷酸微阵列分析技术(ROMA)采用DNA 微阵列检测微生物和哺乳动物基因组中DNA 拷贝数目的改变(图3)[17,18],这些方法也能够检测复制和删除的存在。可是,这些现象确切的本质还不很清楚。扩增区的基因位置不能明确地被标识出来,特别是哺乳动物的基因组,低分辨率的CGH 和ROMA 不能准确地测定断裂点的位置。此外,染色体中基因组外物质的移位和插入不能被完全检测。尽管脉冲场凝胶电泳(PFGE)[19]能够检测到诸如转移等反常现象,但是对哺乳动物的基因组则需要预先知道断裂点及其DNA 印迹;然而低等真核生物能以denono 模式直接检测染色体的异常现象。另一方面,Optical Mapping 系统能够直接分析这些异常的染色体,并且可精确检测和定位基因断裂点的位置,阐明迁移过程??通过成对的单分子限制性图谱或者条形码而非参考基因组。单分子图谱本质上讲是杂交的图谱而非硅片图谱,它们通过序列数据构建而成,这些很像硅片DNA 印迹法,只不过这些探针用于校准或者杂交过程。与所有的限制性图谱一样,已完成的Optical Mapping 的分辨率很容易通过选择限制性内切酶的切割频次来调整。
3 结论
Optical Mapping 正从根本上迅速改变基因分析的方法和结果。该技术的特点??诸如单分子检测、高通量、全基因研究的策略,为基因组学研究开辟了新的“ 战场”。未来的研究工作将集中在人类基因组的Optical
Mapping,以及改进该系统,增加其适应性方面。例如,由于Optical Mapping的底物分子仍具有生物学活性,经探针标记的二级杂交能被利用产生全基因范围基础上的外遗传现象。发展“ 可见测序(Optical Sequencing)”会对单分子Optical Mapping 图3 全基因方法分析基因失常。普通的染色体异常实例以及通过PFG分析、基因微阵列、Optical Mapping 的试验数据。方法产生杠杆作用,从而使分辨率降低到几个碱基对的水平[20,21]。越来越多基因信息的发现,更多的问题也会产生。作者非常乐观地认为Optical Mapping 系统能在将来探索和解决这些问题过程中做出贡献。
Optical Mapping 正从根本上迅速改变基因分析的方法和结果。该技术的特点??诸如单分子检测、高通量、全基因研究的策略,为基因组学研究开辟了新的“ 战场”。未来的研究工作将集中在人类基因组的Optical
Mapping,以及改进该系统,增加其适应性方面。例如,由于Optical Mapping的底物分子仍具有生物学活性,经探针标记的二级杂交能被利用产生全基因范围基础上的外遗传现象。发展“ 可见测序(Optical Sequencing)”会对单分子Optical Mapping 图3 全基因方法分析基因失常。普通的染色体异常实例以及通过PFG分析、基因微阵列、Optical Mapping 的试验数据。方法产生杠杆作用,从而使分辨率降低到几个碱基对的水平[20,21]。越来越多基因信息的发现,更多的问题也会产生。作者非常乐观地认为Optical Mapping 系统能在将来探索和解决这些问题过程中做出贡献。
发布人:2012/9/1 10:45:001336
发布时间:2012/9/1 10:45:00
此新闻已被浏览:1336次
更多相关信息
分类点击排行
- 1500米防水温度记录仪MX2204 HOBO TidbiT MX Tempe 5000
- HOBO无线蓝牙水下温度记录仪MX2203 TidbiT MX
- MX2201新款带蓝牙水下温度记录仪Pendant MX Temp
- 防火报警智能热像Fotric816/Fotric826
- 工程师通用热像仪Fotric 222s/Fotric 222s-1/Fotric 222s-2
- HOBO MX100蓝牙温度记录仪防水等级IP67
- 可程式噪音计TES-1352S带记录可插SD卡
- 数据记录型照度光量子计TES-1339P
- 便携一体式温湿度大气压力计TES-1160/TES-1161
- 高精度水位计MX2001-04-s带无线数据传输


